Aluno: Bruno Rodrigues.
Orientadora: Manoela Kohler.
Trabalho apresentado ao curso BI MASTER como pré-requisito para conclusão de curso e obtenção de crédito na disciplina "Projetos de Sistemas Inteligentes de Apoio à Decisão".
O trabalho de conclusão de curso será a elaboração de uma rede neural para classificação da presença da sequência genética em tumores de cérebro conhecido como MGMT promoter methylation em imagens de ressonância magnética multiparamétrica. Esse problema foi proposto em uma competição hospedada na plataforma Kaggle e que tinha como patrocinadora a Radiological Society of North America (RSNA) que é uma organização sem fins lucrativos.
Atualmente para identificar se um câncer de cérebro tem o MGMT e definir o melhor tratamento, é preciso fazer a extração de uma pequena amostra e fazer a análise genética, o que pode levar várias semanas. Utilizando técnicas de inteligência artificial para determinar se um paciente tem ou não a presença do MGMT no tumor cerebral, os médicos podem tomar decisões de tratamento mais rápido que os métodos atuais e aumentar as chances de cura do paciente.
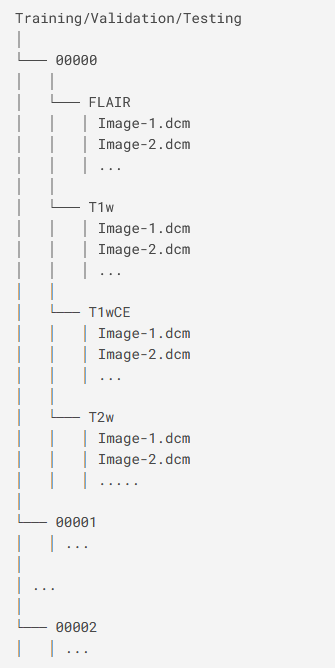
O dataset é da competição da RSNA de Classificação de Tumor Cerebral hospedado no Kaggle é composto por imagens de ressonância magnética multiparamétrica, contendo para cada paciente quatro tipos de imagens, sendo elas a Fluid Attenuated Inversion Recovery (FLAIR), T1-weighted pre-contrast (T1w), T1-weighted post-contrast (T1Gd) e T2-weighted (T2). Para simplificação do problema, o modelo proposto usou como referência cada imagem individual e a classificação de presença do MGMT ou não no paciente, transformando cada arquivo em uma entrada individual da rede neural com sua classificação atribuída.
Foram fornecidas duas pastas, a TRAIN e TEST contendo cada uma a seguinte estrutura:
Dentro das subpastas de cada paciente e tipo de ressonância, temos arquivos do tipo DICOM que é o padrão utilizado na medicina.
Além dos diretórios acima, temos um arquivo CSV que contém a classificação para a presença do MGMT e o identificador de cada paciente. O objetivo é desenvolver um modelo capaz de dizer a probabilidade de cada indivíduo ter o MGMT.
Devido a restrições do ambiente de desenvolvimento do Kaggle, utilizamos apenas parte do dataset neste trabalho.
Nesse trabalho, optamos pela linguagem Python que é amplamente utilizada pela comunidade de ciência de dados. Definimos algumas constantes para melhor organização do código e para armazenar as imagens convertidas em PNG.
# Declaração de constantes
TRAIN_FOLDER = 'train'
TEST_FOLDER = 'test'
mri_types = collections.namedtuple('mri_types', ['FLAIR', 'T1W', 'T1WCE', 'T2W'])
MRI_TYPES = mri_types('FLAIR', 'T1w', 'T1wCE', 'T2w')
PNG_DATASET_DIR = '/kaggle/working/png_dataset'
PNG_TEST_DIR = '/kaggle/working/png_test'
WITH_MGMT_DIR = '/kaggle/working/png_dataset/with_mgmt'
WITHOUT_MGMT_DIR = '/kaggle/working/png_dataset/without_mgmt'Em seguida, criamos três funções auxiliares.
A img_loader(path) é para carregar a imagem DICOM, verificar se é uma imagem vazia e retornar ela no formato de array. Caso o arquivo seja vazio, retorna 0.
A função png_save(path) chama o método acima, verifica se é um array ou 0 e caso seja um array, converte e salva a imagem DICOM no formato PNG.
E imgs_path_finder(folder, patient) percorre todos os pacientes e seus respectivos subdiretórios e retorna o caminho de cada arquivo DICOM de forma ordenada no formato de uma lista.
def img_loader(path):
ds = dcmread(os.path.join(INPUTDIR_PATH, path))
if (ds.pixel_array.sum() != 0):
arr = ds.pixel_array
# arr = tf.constant(arr)
return arr
else:
return 0
def png_save(path):
image = img_loader(path)
if isinstance(image, np.ndarray):
fname = path.replace('/', '-')
fname = fname.replace('dcm', 'png')
imsave(fname, image)
def imgs_path_finder(folder, patient):
images = []
image_path = []
for mri in MRI_TYPES:
images = sorted(os.listdir(os.path.join(folder, patient, mri)),
key=lambda file: int(re.sub('[^0-9]', '', file)))
for img in images:
path = os.path.join(folder,
patient,
mri,
img)
image_path.append(path)
print(path)
return image_pathCriamos então duas variáveis para armazenar a lista de pacientes dos diretórios TRAIN e TEST.
# Lista os pacientes que fazem parte do diretório de treinamento
train_patients = [subdirs for subdirs in os.listdir(TRAIN_FOLDER)]
print('Número de pacientes no diretório de treino', len(train_patients))
# Lista os pacientes que fazem parte do diretório de teste
test_patients = [subdirs for subdirs in os.listdir(TEST_FOLDER)]
print('Número de pacientes no diretório de teste', len(test_patients))
print('Total de pacientes', len(test_patients)+len(train_patients))Número de pacientes no diretório de treino 585
Número de pacientes no diretório de teste 87
Total de pacientes 672
Na descrição da competição, foi informado que os diretórios dos três pacientes 00109, 00123 e 00709 estavam gerando erro no processo de treinamento e orientavam realizar a exclusão deles. A função abaixo serve para eliminar as pastas dos três pacientes.
# Exclusão dos pacientes 00109, 00123, 00709 devido a falha do dataset
patients_delete = ('00109', '00123', '00709')
try:
for patient in patients_delete:
df_label = df_label[df_label.BraTS21ID != patient]
train_patients.remove(patient)
except Exception as err:
print('erro: ', err)
print('Número de pacientes no diretório de treino', len(train_patients))Número de pacientes no diretório de treino 582
Criado a lista contendo uma tuple com o identificador do paciente, o caminho da imagem DICOM e a classificação de presença de MGMT para o diretório TRAIN.
Gerado também a lista com uma tuple com o identificador do paciente e o caminho da imagem DICOM para o diretório TEST. Não foi fornecido no dataset da competição a classificação de MGMT para a pasta TEST.
# retorna uma lista de duas dimensões
# necessário a conversão de patient para list de forma que todos os elementos sejam do tipo list
# acrescenta o label de presença de MGMT
images_path = []
for patient in train_patients[:25]:
images_path.append([
[patient],
imgs_path_finder(TRAIN_FOLDER, patient),
[str(int(df_label[df_label['BraTS21ID']==patient].MGMT_value))]
])
test_images_path = []
for patient in test_patients[:10]:
test_images_path.append([
[patient],
imgs_path_finder(TEST_FOLDER, patient)
])Através do código abaixo, verificamos para cada paciente o Label de classificação para MGMT e chamamos as funções auxiliares para carregar e salvar os arquivos PNG na seguinte estrutura:
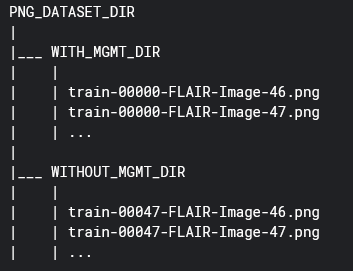
Esse tipo de estrutura foi escolhido para utilizar as facilidades da biblioteca Keras que, a partir desse formato de diretórios, gera automaticamente um dataset para ser utilizado no treinamento dos modelos de rede neural. O Keras entende os subdiretórios WITH_MGMT_DIR e WITHOUT_MGMT_DIR como sendo a categoria e fazendo a associação dessa classe a cada imagem dentro do subdiretório.
for patient in images_path:
if patient[2][0] == '1':
os.chdir(WITH_MGMT_DIR)
for image in patient[1]:
png_save(image)
os.chdir(INPUTDIR_PATH)
else:
os.chdir(WITHOUT_MGMT_DIR)
for image in patient[1]:
png_save(image)
os.chdir(INPUTDIR_PATH)Aqui simplesmente buscamos as imagens de cada paciente no diretório TEST, convertemos em PNG e salvamos na pasta PNG_TEST_DIR.
os.chdir(PNG_TEST_DIR)
for patient in test_images_path:
for image in patient[1]:
png_save(image)
os.chdir(INPUTDIR_PATH)Após especificar o tamanho das nossas imagens e do batch, utilizamos a API do Keras para automaticamente criarmos os datasets de treinamento e validação que iremos utilizar no modelo CNN. Configuramos através dos parâmetros validation_split e subset a porcentagem de 20% para validação e a divisão do diretório de imagens em dois para treino e validação.
image_size = (512, 512)
batch_size = 32
train_ds = tf.keras.preprocessing.image_dataset_from_directory(
PNG_DATASET_DIR,
validation_split=0.2,
subset="training",
seed=1337,
image_size=image_size,
batch_size=batch_size,
)
val_ds = tf.keras.preprocessing.image_dataset_from_directory(
PNG_DATASET_DIR,
validation_split=0.2,
subset="validation",
seed=1337,
image_size=image_size,
batch_size=batch_size,
)Como se tratava de um desafio de classificação de imagens, optamos pela utilização de uma rede neural convolucional, uma vez que esse tipo de rede é o que apresenta os melhores resultados ao se lidar com imagem. Num primeiro momento, elaboramos uma rede simples com apenas quatro camadas de forma experimental.
def make_model(input_shape):
inputs = keras.Input(shape=input_shape)
x = keras.Sequential()
x = layers.experimental.preprocessing.Rescaling(1.0 / 255)(inputs)
x = layers.Conv2D(32, 3, strides=2, padding="same")(x)
x = layers.BatchNormalization()(x)
x = layers.Activation("relu")(x)
x = layers.Conv2D(64, 3, padding="same")(x)
x = layers.BatchNormalization()(x)
x = layers.Activation("relu")(x)
x = layers.GlobalAveragePooling2D()(x)
x = layers.Dropout(0.5)(x)
outputs = layers.Dense(units=1, activation="sigmoid")(x)
return keras.Model(inputs, outputs)
model = make_model(input_shape=image_size + (3,))
model.summary()Model: "model"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
input_1 (InputLayer) [(None, 512, 512, 3)] 0
_________________________________________________________________
rescaling (Rescaling) (None, 512, 512, 3) 0
_________________________________________________________________
conv2d (Conv2D) (None, 256, 256, 32) 896
_________________________________________________________________
batch_normalization (BatchNo (None, 256, 256, 32) 128
_________________________________________________________________
activation (Activation) (None, 256, 256, 32) 0
_________________________________________________________________
conv2d_1 (Conv2D) (None, 256, 256, 64) 18496
_________________________________________________________________
batch_normalization_1 (Batch (None, 256, 256, 64) 256
_________________________________________________________________
activation_1 (Activation) (None, 256, 256, 64) 0
_________________________________________________________________
global_average_pooling2d (Gl (None, 64) 0
_________________________________________________________________
dropout (Dropout) (None, 64) 0
_________________________________________________________________
dense (Dense) (None, 1) 65
=================================================================
Total params: 19,841
Trainable params: 19,649
Non-trainable params: 192
_________________________________________________________________
Definimos então o número de épocas e uma função para salvar o melhor modelo encontrado durante treinamento. Ao compilar o modelo, escolhemos como otimizador o Adam, função de perda do tipo binary_crossentropy, uma vez que nosso problema é do tipo tem ou não MGMT, e como métrica a acurácia.
epochs = 20
callbacks = [
keras.callbacks.ModelCheckpoint(filepath="/kaggle/working/save_at_{epoch}.h5",
save_best_only=True)
]
model.compile(
optimizer=keras.optimizers.Adam(1e-3),
loss="binary_crossentropy",
metrics=["accuracy"],
)
model.fit(
train_ds, epochs=epochs, callbacks=callbacks, validation_data=val_ds,
)Para melhorarmos nossa classificação, optamos na sequência por um modelo pré-treinado utilizando para isso a técnica de transferência de aprendizado. No nosso caso, escolhemos o Xception. O próprio Keras fornece um método para obtermos esse modelo com os pesos pré-treinados no dataset do Imagenet.
Para permitir que o modelo se adeque ao nosso problema, não incluímos a última camada de classificação do Xception ao criarmos nosso modelo base de rede neural.
base_model = keras.applications.Xception(
weights='imagenet', # Load weights pre-trained on ImageNet.
input_shape=(512, 512, 3),
include_top=False) # Do not include the ImageNet classifier at the top.Congelamos então os parâmetros do modelo base para não alterarmos os pesos já pré-treinados e criamos a partir dele um novo modelo, adicionando mais duas camadas, sendo a última a nossa camada de classificação.
base_model.trainable = False
inputs_2 = keras.Input(shape=(512, 512, 3))
x_2 = base_model(inputs_2, training=False)
# Convert features of shape `base_model.output_shape[1:]` to vectors
x_2 = keras.layers.GlobalAveragePooling2D()(x_2)
# A Dense classifier with a single unit (binary classification)
outputs_2 = keras.layers.Dense(1, activation="sigmoid")(x_2)
model_2 = keras.Model(inputs_2, outputs_2)
model_2.summary()Model: "model_1"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
input_3 (InputLayer) [(None, 512, 512, 3)] 0
_________________________________________________________________
xception (Functional) (None, 16, 16, 2048) 20861480
_________________________________________________________________
global_average_pooling2d_1 ( (None, 2048) 0
_________________________________________________________________
dense_1 (Dense) (None, 1) 2049
=================================================================
Total params: 20,863,529
Trainable params: 2,049
Non-trainable params: 20,861,480
_________________________________________________________________
E assim como a primeira rede, fazemos o treinamento dela.
epochs = 20
callbacks = [
keras.callbacks.ModelCheckpoint(filepath="/kaggle/working/transfer_save_at_{epoch}.h5",
save_best_only=True)
]
model_2.compile(optimizer=keras.optimizers.Adam(),
loss=keras.losses.BinaryCrossentropy(from_logits=True),
metrics=[keras.metrics.BinaryAccuracy()])
model_2.fit(train_ds, epochs=epochs, callbacks=callbacks, validation_data=val_ds)Ao treinarmos o nosso primeiro modelo e observando a saída do método fit, podemos perceber que a nossa rede não performa adequadamente, inclusive ela aparentemente não faz nenhum tipo de aprendizado significativo após a segunda época.
Resultado do primeiro modelo
Epoch 1/20
308/308 [==============================] - 81s 235ms/step - loss: 0.6922 - accuracy: 0.5740 - val_loss: 0.7563 - val_accuracy: 0.3718
Epoch 2/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6793 - accuracy: 0.6046 - val_loss: 0.6582 - val_accuracy: 0.6282
Epoch 3/20
308/308 [==============================] - 77s 245ms/step - loss: 0.6749 - accuracy: 0.5988 - val_loss: 0.6518 - val_accuracy: 0.6233
Epoch 4/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6675 - accuracy: 0.6103 - val_loss: 0.6553 - val_accuracy: 0.6282
Epoch 5/20
308/308 [==============================] - 77s 244ms/step - loss: 0.6656 - accuracy: 0.6136 - val_loss: 0.6552 - val_accuracy: 0.6282
Epoch 6/20
308/308 [==============================] - 77s 246ms/step - loss: 0.6673 - accuracy: 0.6112 - val_loss: 0.6522 - val_accuracy: 0.6249
Epoch 7/20
308/308 [==============================] - 78s 248ms/step - loss: 0.6648 - accuracy: 0.6168 - val_loss: 0.6565 - val_accuracy: 0.6282
Epoch 8/20
308/308 [==============================] - 77s 245ms/step - loss: 0.6646 - accuracy: 0.6133 - val_loss: 0.6531 - val_accuracy: 0.6282
Epoch 9/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6658 - accuracy: 0.6126 - val_loss: 0.6527 - val_accuracy: 0.6282
Epoch 10/20
308/308 [==============================] - 78s 249ms/step - loss: 0.6654 - accuracy: 0.6151 - val_loss: 0.6548 - val_accuracy: 0.6282
Epoch 11/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6640 - accuracy: 0.6112 - val_loss: 0.6531 - val_accuracy: 0.6282
Epoch 12/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6630 - accuracy: 0.6142 - val_loss: 0.6540 - val_accuracy: 0.6282
Epoch 13/20
308/308 [==============================] - 78s 248ms/step - loss: 0.6639 - accuracy: 0.6163 - val_loss: 0.6542 - val_accuracy: 0.6273
Epoch 14/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6631 - accuracy: 0.6152 - val_loss: 0.6542 - val_accuracy: 0.6282
Epoch 15/20
308/308 [==============================] - 77s 247ms/step - loss: 0.6624 - accuracy: 0.6139 - val_loss: 0.6531 - val_accuracy: 0.6282
Epoch 16/20
308/308 [==============================] - 78s 250ms/step - loss: 0.6630 - accuracy: 0.6146 - val_loss: 0.6543 - val_accuracy: 0.6282
Epoch 17/20
308/308 [==============================] - 77s 246ms/step - loss: 0.6629 - accuracy: 0.6183 - val_loss: 0.6531 - val_accuracy: 0.6282
Epoch 18/20
308/308 [==============================] - 78s 250ms/step - loss: 0.6640 - accuracy: 0.6133 - val_loss: 0.6533 - val_accuracy: 0.6282
Epoch 19/20
308/308 [==============================] - 78s 249ms/step - loss: 0.6633 - accuracy: 0.6140 - val_loss: 0.6526 - val_accuracy: 0.6282
Epoch 20/20
308/308 [==============================] - 78s 248ms/step - loss: 0.6648 - accuracy: 0.6111 - val_loss: 0.6525 - val_accuracy: 0.6290
<tensorflow.python.keras.callbacks.History at 0x7f514bc06110>
Já o modelo dois, que tem como base o Xception, obtemos uma acurácia próxima de 80%.
Resultado do segundo modelo
Epoch 1/20
308/308 [==============================] - 152s 478ms/step - loss: 0.6826 - binary_accuracy: 0.6167 - val_loss: 0.5724 - val_binary_accuracy: 0.6908
Epoch 2/20
308/308 [==============================] - 146s 472ms/step - loss: 0.6004 - binary_accuracy: 0.6893 - val_loss: 0.5349 - val_binary_accuracy: 0.7323
Epoch 3/20
308/308 [==============================] - 146s 472ms/step - loss: 0.5515 - binary_accuracy: 0.7122 - val_loss: 0.5093 - val_binary_accuracy: 0.7482
Epoch 4/20
308/308 [==============================] - 146s 472ms/step - loss: 0.5214 - binary_accuracy: 0.7281 - val_loss: 0.5214 - val_binary_accuracy: 0.7347
Epoch 5/20
308/308 [==============================] - 146s 472ms/step - loss: 0.5127 - binary_accuracy: 0.7476 - val_loss: 0.4891 - val_binary_accuracy: 0.7624
Epoch 6/20
308/308 [==============================] - 147s 473ms/step - loss: 0.4894 - binary_accuracy: 0.7540 - val_loss: 0.5035 - val_binary_accuracy: 0.7531
Epoch 7/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4829 - binary_accuracy: 0.7640 - val_loss: 0.4712 - val_binary_accuracy: 0.7710
Epoch 8/20
308/308 [==============================] - 147s 472ms/step - loss: 0.4746 - binary_accuracy: 0.7698 - val_loss: 0.4639 - val_binary_accuracy: 0.7693
Epoch 9/20
308/308 [==============================] - 147s 472ms/step - loss: 0.4652 - binary_accuracy: 0.7762 - val_loss: 0.4589 - val_binary_accuracy: 0.7799
Epoch 10/20
308/308 [==============================] - 146s 472ms/step - loss: 0.4549 - binary_accuracy: 0.7860 - val_loss: 0.4715 - val_binary_accuracy: 0.7669
Epoch 11/20
308/308 [==============================] - 146s 472ms/step - loss: 0.4544 - binary_accuracy: 0.7874 - val_loss: 0.4609 - val_binary_accuracy: 0.7730
Epoch 12/20
308/308 [==============================] - 146s 470ms/step - loss: 0.4442 - binary_accuracy: 0.7948 - val_loss: 0.4519 - val_binary_accuracy: 0.7799
Epoch 13/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4393 - binary_accuracy: 0.7957 - val_loss: 0.4516 - val_binary_accuracy: 0.7795
Epoch 14/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4329 - binary_accuracy: 0.7968 - val_loss: 0.4439 - val_binary_accuracy: 0.7884
Epoch 15/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4285 - binary_accuracy: 0.8024 - val_loss: 0.4667 - val_binary_accuracy: 0.7685
Epoch 16/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4254 - binary_accuracy: 0.8059 - val_loss: 0.4350 - val_binary_accuracy: 0.7958
Epoch 17/20
308/308 [==============================] - 147s 472ms/step - loss: 0.4189 - binary_accuracy: 0.8115 - val_loss: 0.4641 - val_binary_accuracy: 0.7811
Epoch 18/20
308/308 [==============================] - 146s 470ms/step - loss: 0.4204 - binary_accuracy: 0.8051 - val_loss: 0.4326 - val_binary_accuracy: 0.7962
Epoch 19/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4164 - binary_accuracy: 0.8066 - val_loss: 0.4320 - val_binary_accuracy: 0.7905
Epoch 20/20
308/308 [==============================] - 146s 471ms/step - loss: 0.4107 - binary_accuracy: 0.8124 - val_loss: 0.4281 - val_binary_accuracy: 0.7978
<tensorflow.python.keras.callbacks.History at 0x7f512c1f4c10>
Finalizado o treinamento, os modelos foram salvos em arquivos do formato .h5 para posteriormente serem restaurados e utilizados para fazer as predições das imagens de teste. Para fins desse trabalho, utilizamos diretamente o modelo treinado sem restaurarmos de arquivo.
Ao aplicar nosso segundo modelo, obtemos as probalidades de cada imagem ter presença de MGMT ou não.
#restored_model = keras.models.load_model("/kaggle/input/saved-models/transfer_save_at_17.h5")
#restored_model.summary()
predictions = []
i = 0
for file in os.listdir(PNG_TEST_DIR)[:100]:
image = tf.keras.preprocessing.image.load_img(os.path.join(PNG_TEST_DIR, file),
target_size=image_size)
input_arr = keras.preprocessing.image.img_to_array(image)
input_arr = np.array([input_arr]) # Convert single image to a batch.
predictions.append([file, model_2.predict(input_arr)])
predictions[['test-00082-FLAIR-Image-113.png', array([[0.05473313]], dtype=float32)],
['test-00013-T2w-Image-269.png', array([[0.647184]], dtype=float32)],
['test-00079-T2w-Image-281.png', array([[0.21394956]], dtype=float32)],
['test-00015-T1wCE-Image-163.png', array([[0.17550711]], dtype=float32)],
['test-00080-FLAIR-Image-306.png', array([[0.44676653]], dtype=float32)],
['test-00082-FLAIR-Image-295.png', array([[0.7266254]], dtype=float32)],
['test-00013-T2w-Image-83.png', array([[0.44098884]], dtype=float32)],
['test-00013-FLAIR-Image-424.png', array([[0.3264906]], dtype=float32)],
['test-00082-T1w-Image-9.png', array([[0.6909217]], dtype=float32)],
['test-00047-FLAIR-Image-28.png', array([[0.24077713]], dtype=float32)],
['test-00091-FLAIR-Image-326.png', array([[0.15971619]], dtype=float32)],
['test-00047-FLAIR-Image-150.png', array([[0.20528059]], dtype=float32)],
['test-00080-T2w-Image-87.png', array([[0.91539294]], dtype=float32)],
['test-00080-T2w-Image-144.png', array([[0.78349453]], dtype=float32)],
['test-00001-T2w-Image-354.png', array([[0.00290455]], dtype=float32)],
['test-00027-T1wCE-Image-103.png', array([[0.11121766]], dtype=float32)],
['test-00082-T2w-Image-124.png', array([[0.537534]], dtype=float32)],
['test-00091-FLAIR-Image-316.png', array([[0.16906482]], dtype=float32)],
['test-00015-T1wCE-Image-91.png', array([[0.33880532]], dtype=float32)],
['test-00079-T2w-Image-79.png', array([[0.43134928]], dtype=float32)],
['test-00015-FLAIR-Image-482.png', array([[0.49448198]], dtype=float32)],
Ao elaborar esse trabalho, podemos verificar uma aplicação de deep learning com grande impacto benéfico em pacientes com tumores cerebrais, melhorando assim suas chances de cura e sobrevida.
Vimos também que ao utilizar bibliotecas como o Tensorflow e Keras facilitam o trabalho de desenvolvimento, uma vez que eles já nos trazem ferramentas prontas para uso na resolução dos nossos problemas.
Um ponto a melhorar no futuro seria realizar um ajuste fino do segundo modelo, transformando os parâmetros internos da base em treináveis e fazer um novo treinamento com poucas épocas e um otimizador com ganho pequeno para dessa forma aumentarmos a acurácia final.
Matrícula: 192.671.026
Pontifícia Universidade Católica do Rio de Janeiro
Curso de Pós Graduação Business Intelligence Master